やること
人生で一度は進化系統樹を描いてみたいですよね。本来、系統樹は特定の遺伝子の塩基配列の相同性から作られるものですが、どんなものでも距離行列があれば描けます。今回は、PythonのBio.Phyloを使用してポケモンの系統樹を描いてみましょう。

・・・おや!? ○○のようすが・・・!
神戸大学の生物学の講義で痛烈に指摘されていますが、キャタピー→トランセル→バタフリーと変化することを、生物学的には「進化」とは呼びません。正しくは「変態」です。
ここでは、ポケモンの種族値のバランスだけを考慮し、「ポケモンたちが何らかの祖先から(生物学的な)進化をしてきた」と仮定して系統樹を描くことにします。ゲーム内の進化とは関係ありませんし、ポケモンたちのタイプや見た目も関係ありません。
実行環境
WinPython3.6をおすすめしています。
参考文献
Phyloのマニュアルには塩基配列を入力とする操作しか書かれておらず、独自の距離行列から系統樹を描く方法がなかなか見つかりませんでした。しかし、同じようなことをしようとしている奇特な方を見つけました。
ポケモンのデータベース
例によって、ポケモンのデータベースはこちらを使わせていただきました。
2行ほど変な箇所があるので、手動で修正しました。
pip, import
Phyloを入れます。
pip install biopythonまた、グラフ中で日本語を表示したいので、こちらもpipします。
pip install japanize-matplotlib今回使うパッケージをインポートします。
import numpy as np
import matplotlib.pyplot as plt
import japanize_matplotlib
from Bio.Phylo import TreeConstruction, draw
from Bio.Phylo.TreeConstruction import DistanceTreeConstructor全ポケモン名の読み込み
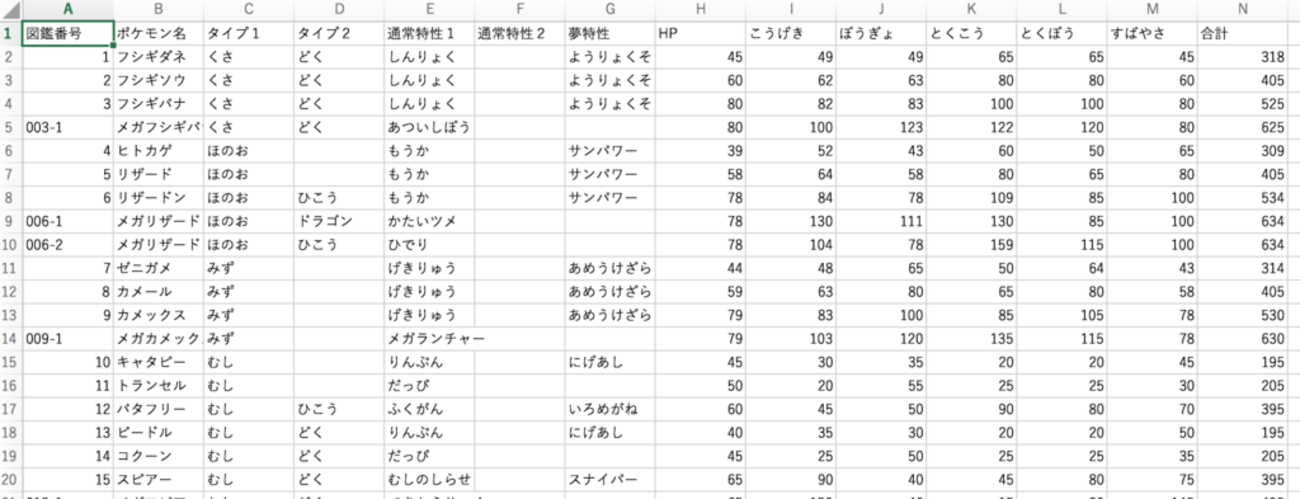
「pokemon_status.csv」の全行について、名前(2列目)と種族値(8~13列目)を読み込みます。ただし全ポケモンだと多いので、最初の151匹(メガ・アローラを含めると184匹)に削減しておきます。
#============================
#全ポケモンデータの読み込み
#============================
#ファイル名
file = open('pokemon_status.csv', 'r')
#
name = []
data = []
#1行目はラベルなのでカラ読み
file.readline()
#2行目以降、ポケモン名と種族値を読み込む
while 1:
tmp = file.readline().split(',')
if len(tmp) > 1:
name.append(tmp[1])
data.append([float(tmp[7]), float(tmp[8]), float(tmp[9]), float(tmp[10]), float(tmp[11]), float(tmp[12])])
else:
break
file.close()
data = np.array(data)
print('ポケモンの数\n{}'.format(len(name)))
print('最初の4匹の名前\n{}'.format(name[:4]))
print('最初の4匹の種族値\n{}'.format(data[:4]))
print('ヒマナッツの種族値\n{}'.format(data[224]))
#ポケモンを151匹に減らす
name = name[:184]
data = data[:184]
print('削減後、最後のポケモン名\n{}'.format(name[-1]))ポケモンの数
909
最初の4匹の名前
['フシギダネ', 'フシギソウ', 'フシギバナ', 'メガフシギバナ']
最初の4匹の種族値
[[ 45. 49. 49. 65. 65. 45.]
[ 60. 62. 63. 80. 80. 60.]
[ 80. 82. 83. 100. 100. 80.]
[ 80. 100. 123. 122. 120. 80.]]
ヒマナッツの種族値
[30. 30. 30. 30. 30. 30.]
削減後、最後のポケモン名
ミュウメガやアローラを含めるとポケモン数は909匹でした。種族値は左からHP、こうげき、ぼうぎょ、とくこう、とくぼう、すばやさです。ヒマナッツは弱いです。
種族値の規格化
種族値の合計が100になるように規格化します。こうすることで、種族値の絶対値ではなく、バランス(比率)を考慮した系統樹ができます。
#============================
#種族値を合計100に規格化する
#============================
for i in range(len(data)):
data[i] = data[i] / np.sum(data[i]) * 100
print('最初の4匹の種族値\n{}'.format(data[:4]))最初の4匹の種族値
[[14.1509434 15.40880503 15.40880503 20.44025157 20.44025157 14.1509434 ]
[14.81481481 15.30864198 15.55555556 19.75308642 19.75308642 14.81481481]
[15.23809524 15.61904762 15.80952381 19.04761905 19.04761905 15.23809524]
[12.8 16. 19.68 19.52 19.2 12.8 ]]距離行列の作成
ポケモン同士の距離を計算し、下三角行列で表します。距離は種族値の6次元ユークリッド距離とします。909匹だと909C2=412,686回の計算、184匹だと184C2=16,836回の計算ですが、一瞬で済みます。
#============================
#距離行列の作成
#============================
#下三角行列の作成
dm = np.zeros((len(name), len(name)))
for i in range(len(name)):
for j in range(i, len(name)):
dm[j, i] = np.sum((data[i] - data[j])**2)**0.5
print('最初の4匹の距離行列\n{}'.format(dm[0:4, 0:4]))
#リスト形式にしないとPhyloに入れられないのでこれも用意する
dm_list = []
for i in range(len(name)):
dm_list.append(list(dm[i, :i+1]))
print('最初の4匹の距離行列\n{}'.format(dm_list[0:4]))最初の4匹の距離行列
[[0. 0. 0. 0. ]
[1.36286858 0. 0. 0. ]
[2.5391783 1.2306716 0. 0. ]
[4.96264053 5.09590122 5.22117718 0. ]]
最初の4匹の距離行列
[[0.0], [1.3628685808992327, 0.0], [2.5391782995787513, 1.2306716042247077, 0.0], [4.962640525857675, 5.095901219254242, 5.221177181102225, 0.0]]ちょっと面倒なことに、Phyloは上記のdm_listの形式でしか入力を受け付けません。
近隣結合法で系統樹を描く
文献が少なくてちょっと苦労しました。詳細はコメントを参照してください。
#============================
#Neighbor Joining法
#============================
#Distance matrix型の用意
DM = TreeConstruction._DistanceMatrix(name, dm_list)
#nj木に変換
tree = DistanceTreeConstructor().nj(DM)
#クレード名か'Inner#'のものを''に改名する(見づらいので)
for clade in tree.find_clades():
if 'Inner' in clade.name:
clade.name = ''
#線の色
#tree.root.color = 'gray'
#highlight = tree.common_ancestor('フリーザー', 'サンダー', 'ファイヤー')
#highlight.color = 'magenta'
#表示
#draw(tree) だけでも表示可能だが、グラフサイズを大きくしたい
fig = plt.figure(figsize=(30, 30), dpi=150)
axes = fig.add_subplot(1, 1, 1)
draw(tree, axes=axes)
#plt.savefig('aaa.png')
plt.show()ミュウまで184匹の系統樹です、ご査収ください。
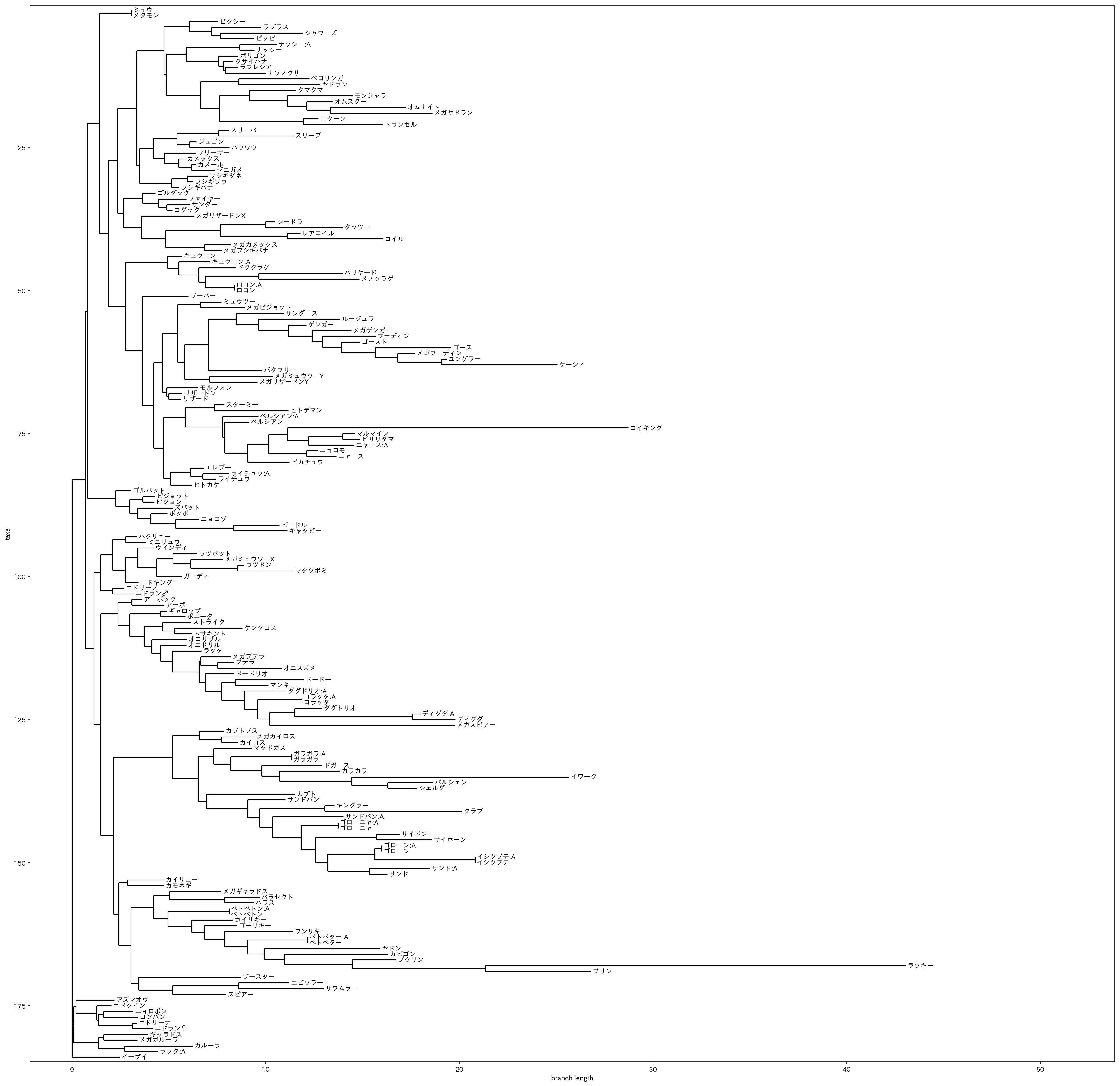
太古の昔、ポケモンは「イーブイ」「アズマオウのなかま」「その他」に分かれました。イーブイは太古からその姿を変えることなく現代まで来ました※1。その秘めたる力が「進化の石」で発揮されるのでしょう。また、一人だけ圧倒的に進化している宇宙人がいます。「ピンクの悪魔」ことラッキーですね。
眺めているだけで一日が過ぎていきそうです。
※1専門家にご指摘いただきました。このような解釈は正しくありませんので、あくまで妄想とお考えください。
伝説のポケモンはいつ伝説となったのか
#線の色
tree.root.color = 'gray'
highlight = tree.common_ancestor('フリーザー', 'サンダー', 'ファイヤー')
highlight.color = 'magenta'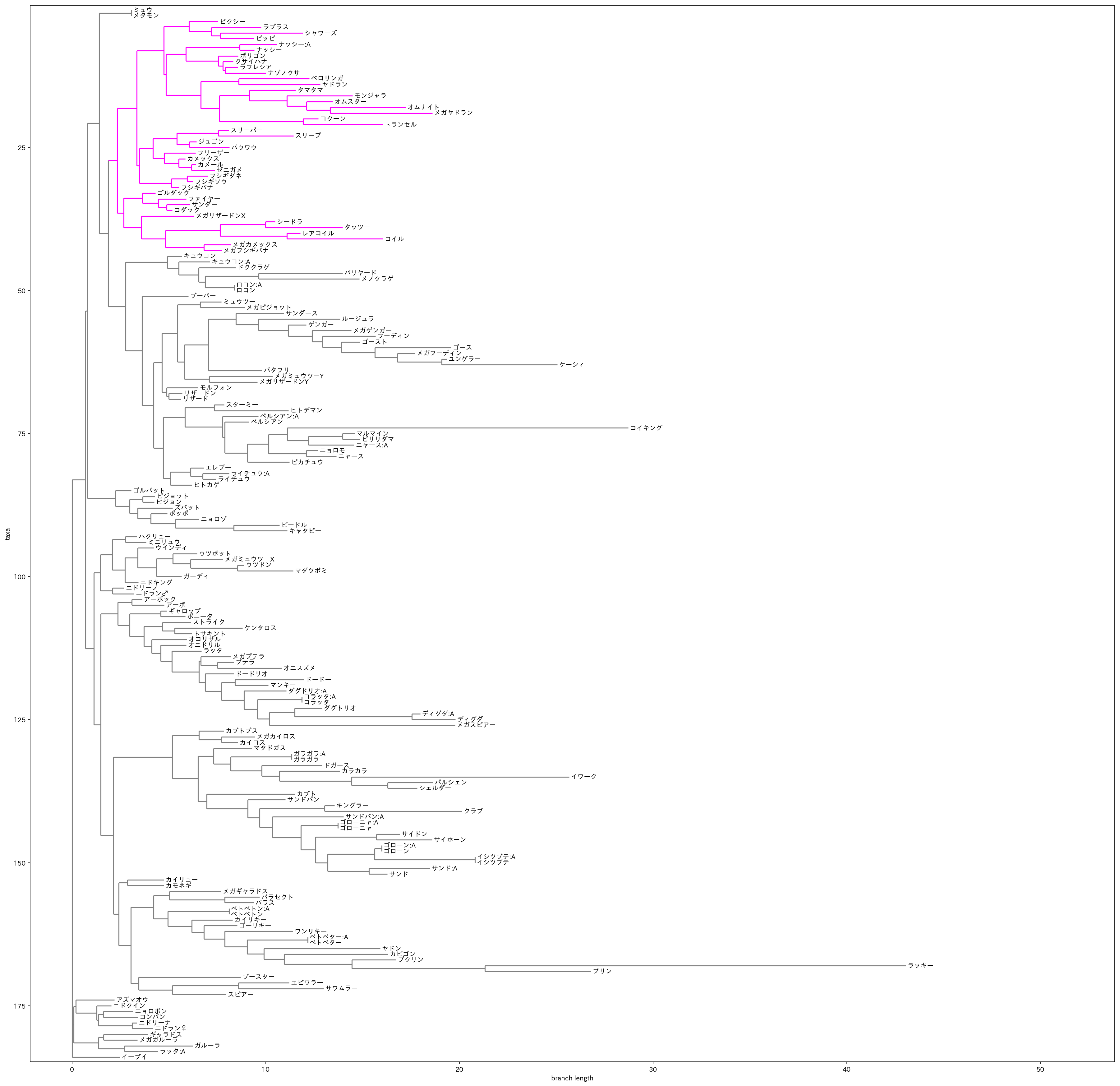
いや、ちょっと待ってください?
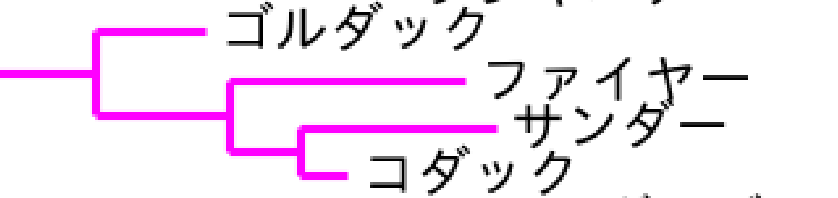
909匹の系統樹
描画に10分くらいかかりました…。画像をクリックすると高解像度にジャンプできますが、1.6MBくらいあるので注意してください。
ラッキーよりもさらに進化している問題児がいました(結論)。
【追記】
専門家にご意見を頂きました。現状はただの階層クラスタリングなので、ポケモンの特徴を遺伝子で表現したほうが、良い考察が得られると思います。