はじめに
vcoptとは
遺伝的アルゴリズム(Genetic Algorithm, GA)による本格的な組合せ最適化がお手軽にできる無料のPythonパッケージです。読み方は「ぶいしーおぷと」です。
設計理念
GAは「調整が難しい」と言われること多いですが、vcoptではすべてのハイパーパラメータが自動的かつ動的に決定されるので、思考停止で使うことができます。
- 高い汎用性(さまざまな最適化問題に対応できる)
- 高いユーザビリティ(ユーザーがハイパーパラメータを設定する必要がない)
- 高い計算効率(並列計算が可能な有償版もある)
できること
| <目的> | <コマンド> |
| 順序(並び替え)の局所最適化 | vcopt().opt2() |
| 順序(並び替え)の大域最適化(*1) | vcopt().tspGA() |
| 離散値の組合せ最適化(*1) | vcopt().dcGA() |
| 選択肢の組合せ最適化(*1) | vcopt().setGA() |
| 連続値の組合せ最適化(*1) | vcopt().rcGA() |
(*1) いずれも大域解を保証するものではありません
インストール
実行環境
Python3で使うことができますが、一応3.6をおすすめしています。Google colaboratoryでも使用可能です。
WinPython3.6をおすすめしています。
Google Colaboratoryが利用可能です。

pip
特に理由がない限り、最新バージョンを使用してください。
pip install vcoptimport
from vcopt import vcopt順序の局所最適化|vcopt().opt2()
このコマンドでは、巡回セールスマン問題に代表される、順序の局所最適化ができます。2-opt法を独自に高速化したアルゴリズムを用いています。
使い方
#2-opt法で最適化
para, score = vcopt().opt2(para,
score_func,
aim,
show_para_func=None,
seed=None,
step_max=float('inf'))| para | 必須 | list, numpy.ndarray | 並び替えたい要素の1次元配列です。 要素は int, float, str のいずれでも良いですが、例えば [2, ‘a’] のように混在する場合には [‘2’, ‘a’] のようにすべて str として認識されます。配列は list でも numpy.ndarray でも良いですが、内部で numpy.ndarray に変換されます。 <例1> [0, 1, 2, 3, 4] <例2> [-10, -1, 0, 1, 10] <例3> [‘v’, ‘c’, ‘o’, ‘p’, ‘t’] |
| score_func | 必須 | 関数 | 評価関数です。para を受け取って評価値(単一の数値)を返す関数を設定してください。 詳細は共通の説明をご覧ください。 |
| aim | 必須 | int, float | 目標評価値です。 <例1> 0.0 <例2> 999999 <例3> -999999 |
| show_para_func | 任意 | 関数 | para を可視化する関数です。ステップ毎に、並び替え途中の para を受け取って可視化できます。 詳細は参考記事をご覧ください。 <受け取る para の例1> [3, 2, 0, 4, 1] <受け取る para の例2> [1, -10, 10, -1, 0] <受け取る para の例3> [‘o’, ”c, ‘v’, ‘t’, ‘p’] |
| seed | 任意 | int | 乱数シードを指定できます。 |
| step_max | 任意 | int | 最大処理ステップ数を指定できます。 |
具体的な使用例はチュートリアルを参考にしてください。
順序の大域最適化|vcopt().tspGA()
このコマンドでは、巡回セールスマン問題に代表される、順序の大域最適化ができます。親のもつ「辺」を受け継ぐような交叉法を採用し、独自の高速化アルゴリズムを用いています。
ハイパーパラメータ一覧
| 個体数、親数、子数 | 10n(*2), 2, 4 |
| 交叉法 | 親のもつ辺を、2手先までを重み付けして確率的に採用する |
| 突然変異率 | 各辺ともおおむね1/n |
| 選択法 | エリート選択 |
| 終了条件 | 目標達成もしくは個体群の平均評価値に変化がなくなったら |
(*2) nは次元数
使い方
#GAで最適化
para, score = vcopt().tspGA(para_range,
score_func,
aim,
show_pool_func='bar',
seed=None,
pool_num=None,
max_gen=None,
core_num=1)| para_range | 必須 | list, numpy.ndarray | 並び替えたい要素の1次元配列です。 要素は int, float, str のいずれでも良いですが、例えば [1, ‘a’] のように混在する場合には [‘1’, ‘a’] のようにすべて str として認識されます。配列は list でも numpy.ndarray でも良いですが、内部で numpy.ndarray に変換されます。 <例1> [0, 1, 2, 3, 4] <例2> [-10, -1, 0, 1, 10] <例3> [‘v’, ‘c’, ‘o’, ‘p’, ‘t’] |
| score_func | 必須 | 関数 | 評価関数です。para を受け取って評価値(単一の数値)を返す関数を設定してください。 詳細は共通の説明をご覧ください。 |
| aim | 必須 | int, float, str | 目標評価値です。比較演算子を含む文字列も指定可能です。 <例1> 0.0 <例2>(とにかく大きくしたいとき) 999999 <例3> ‘<=0.2’ ‘>1000’ |
| show_pool_func | 任意 | 関数 | pool を可視化する関数です。ステップ毎に、pool を受け取って可視化できます。None, ‘bar’, ‘print’, ‘plot’ から選べ、またplotを保存する ‘(path)/’ を指定したり、独自に定義することもできます。詳細は共通の説明をご覧ください。 |
| seed | 任意 | int | 乱数シードを指定できます。 |
| pool_num | 任意 | int | 個体数を指定できます。必ず2以上の偶数としてください。 |
| max_gen | 任意 | int | 最大世代数を指定できます。一定の世代数(計算時間)で終了したい場合に有効です。 |
| core_num | 任意 | int | 並列計算用のノード数を指定できます。※利用にはパスワードが必要です。→有償版「vc-grendle」 |
具体的な使用例は次の記事を参考にしてください。
離散値の組合せ最適化|vcopt().dcGA()
このコマンドでは、モンタージュで文章を完成させる、といった離散値の組合せ最適化ができます。二点交叉と一様交叉を組み合わせた交叉法を採用しています。
ハイパーパラメータ一覧
| 個体数、親数、子数 | 10n(*2), 2, 4 |
| 交叉法 | 二点交叉と一様交叉を組み合わせた方法 |
| 突然変異率 | 各遺伝子ともおおむね1/n |
| 選択法 | エリート選択 |
| 終了条件 | 目標達成もしくは個体群の平均評価値に変化がなくなったら |
(*2) nは次元数
使い方
#GAで最適化
para, score = vcopt().dcGA(para_range,
score_func,
aim,
show_pool_func=None,
seed=None,
pool_num=None,
max_gen=None,
core_num=1)| para_range | 必須 | list, numpy.ndarray | 取りうる遺伝子の選択肢を列挙した2次元配列です。ただし遺伝子が1つしかない場合は1次元配列でも構いません。 要素は int, float, str のいずれでも良いですが、例えば [[1, ‘a’]] のように混在する場合には [[‘1’, ‘a’]] のようにすべて str として認識されます。配列は list でも numpy.ndarray でも良いですが、内部で numpy.ndarray に変換されます。 <例0> [0, 1, 2, 3, 4] <例1> [[0, 1, 2, 3, 4], [0, 1, 2, 3, 4], [0, 1, 2, 3, 4]] <例2> [[0, 1, 2, 3, 4], [5, 6, 7, 8, 9], [-10, -1, 0, 1, 10]] <例3> [[‘私は’, ‘僕は’, ‘俺は’, ‘吾輩は’, ‘拙者は’], [0, 1, 10, 100, 1000], [‘歳の’, ‘匹の’, ‘戦錬磨の’] [‘猫でございます’, ‘猫です’, ‘猫だ’, ‘猫である’, ‘猫どすえ’]] |
| score_func | 必須 | 関数 | 評価関数です。para を受け取って評価値(単一の数値)を返す関数を設定してください。 詳細は共通の説明をご覧ください。 |
| aim | 必須 | int, float, str | 目標評価値です。比較演算子を含む文字列も指定可能です。 <例1> 0.0 <例2>(とにかく大きくしたいとき) 999999 <例3> ‘<=0.2’ ‘>1000’ |
| show_pool_func | 任意 | 関数 | pool を可視化する関数です。ステップ毎に、pool を受け取って可視化できます。None, ‘bar’, ‘print’, ‘plot’ から選べ、またplotを保存する ‘(path)/’ を指定したり、独自に定義することもできます。詳細は共通の説明をご覧ください。 |
| seed | 任意 | int | 乱数シードを指定できます。 |
| pool_num | 任意 | int | 個体数を指定できます。必ず2以上の偶数としてください。 |
| max_gen | 任意 | int | 最大世代数を指定できます。一定の世代数(計算時間)で終了したい場合に有効です。 |
| core_num | 任意 | int | 並列計算用のノード数を指定できます。※利用にはパスワードが必要です。→有償版「vc-grendle」 |
具体的な使用例は次の記事を参考にしてください。
選択肢の組合せ最適化|vcopt().setGA()
このコマンドでは、100個の選択肢の中から重複せずに10個を選ぶ、といった選択肢の組合せ最適化ができます。親の持つ共通集合を引き継ぐ交叉法を採用しています。
ハイパーパラメータ一覧
| 個体数、親数、子数 | 10n(*2), 2, 4 |
| 交叉法 | 親の持つ共通集合を引き継ぐ方法 |
| 突然変異率 | 各遺伝子ともおおむね1/n |
| 選択法 | エリート選択 |
| 終了条件 | 目標達成もしくは個体群の平均評価値に変化がなくなったら |
(*2) nは次元数(ここではset_num)
使い方
#GAで最適化
para, score = vcopt().setGA(para_range,
set_num,
score_func,
aim,
show_pool_func=None,
seed=None,
pool_num=None,
max_gen=None,
core_num=1)| para_range | 必須 | list, numpy.ndarray | 取りうる遺伝子の選択肢を列挙した1次元配列です。 要素は int, float, str のいずれでも良いですが、例えば [1, ‘a’] のように混在する場合には [‘1’, ‘a’] のようにすべて str として認識されます。配列は list でも numpy.ndarray でも良いですが、内部で numpy.ndarray に変換されます。 <例1> [0, 1, 2, 3, 4] <例2> [-10, -1, 0, 1, 10] <例3> [‘v’, ‘c’, ‘o’, ‘p’, ‘t’] |
| set_num | 必須 | int | para_rangeから何個選ぶかを指定します。 <例> 3 |
| score_func | 必須 | 関数 | 評価関数です。para を受け取って評価値(単一の数値)を返す関数を設定してください。paraはソートされた状態で渡されます(要確認)。 詳細は共通の説明をご覧ください。 |
| aim | 必須 | int, float, str | 目標評価値です。比較演算子を含む文字列も指定可能です。 <例1> 0.0 <例2>(とにかく大きくしたいとき) 999999 <例3> ‘<=0.2’ ‘>1000’ |
| show_pool_func | 任意 | 関数 | pool を可視化する関数です。ステップ毎に、pool を受け取って可視化できます。None, ‘bar’, ‘print’, ‘plot’ から選べ、またplotを保存する ‘(path)/’ を指定したり、独自に定義することもできます。詳細は共通の説明をご覧ください。 |
| seed | 任意 | int | 乱数シードを指定できます。 |
| pool_num | 任意 | int | 個体数を指定できます。必ず2以上の偶数としてください。 |
| max_gen | 任意 | int | 最大世代数を指定できます。一定の世代数(計算時間)で終了したい場合に有効です。 |
| core_num | 任意 | int | 並列計算用のノード数を指定できます。※利用にはパスワードが必要です。→有償版「vc-grendle」 |
具体的な使用例は次の記事を参考にしてください。
連続値の組合せ最適化|vcopt().rcGA()
このコマンドでは連続値の組合せ最適化ができます。交叉法には REX (Real-Coded Ensemble Crossover) を採用しています。
ハイパーパラメータ一覧
| 個体数、親数、子数 | 10n(*2), 2, 4 |
| 交叉法 | REX (Real-Coded Ensemble Crossover) |
| 突然変異率 | (REXに内包) |
| 選択法 | エリート選択 |
| 終了条件 | 目標達成もしくは個体群がもつ各遺伝子の分散が十分に小さくなったら |
(*2) nは次元数
使い方
#GAで最適化
para, score = vcopt().rcGA(para_range,
score_func,
aim,
show_pool_func=None,
seed=None,
pool_num=None,
max_gen=None,
core_num=1)| para_range | 必須 | list, numpy.ndarray | 各遺伝子が取りうる実数値の最小値と最大値を列挙した2次元配列です。ただし遺伝子が1つしかない場合は1次元配列でも構いません。 要素は int, float のいずれかです。配列は list でも numpy.ndarray でも良いですが、内部で numpy.ndarray に変換されます。 <例0> [-5, 5] <例1> [[-5.12, -5.12], [-5.12, -5.12], [-5.12, -5.12], [-5.12, -5.12]] <例2> [[0, 1], [0, 100], [-10, 0], [-100, 0]] |
| score_func | 必須 | 関数 | 評価関数です。para を受け取って評価値(単一の数値)を返す関数を設定してください。 詳細は共通の説明をご覧ください。 |
| aim | 必須 | int, float, str | 目標評価値です。比較演算子を含む文字列も指定可能です。 <例1> 0.0 <例2>(とにかく大きくしたいとき) 999999 <例3> ‘<=0.2’ ‘>1000’ |
| show_pool_func | 任意 | 関数 | pool を可視化する関数です。ステップ毎に、pool を受け取って可視化できます。None, ‘bar’, ‘print’, ‘plot’ から選べ、またplotを保存する ‘(path)/’ を指定したり、独自に定義することもできます。詳細は共通の説明をご覧ください。 |
| seed | 任意 | int | 乱数シードを指定できます。 |
| pool_num | 任意 | int | 個体数を指定できます。必ずnの倍数(*2)としてください。 |
| max_gen | 任意 | int | 最大世代数を指定できます。一定の世代数(計算時間)で終了したい場合に有効です。 |
| core_num | 任意 | int | 並列計算用のノード数を指定できます。※利用にはパスワードが必要です。→有償版「vc-grendle」 |
(*2) nは次元数、すなわち len(para_range) です
具体的な使用例は次の記事を参考にしてください。
役に立つTips
評価関数の書き方|score_func()
score_func() は評価値を計算する必須関数で、score_func=your_func のように渡します。opt2()および3種のGAの最中に頻繁に呼び出されます。
次のルールに従って書きます。
#評価関数
def score_func(para):
#paraを使ってスコアを計算する
score = ...
#スコアを返す
return scorepara は次のような1次元配列です(ndarray型)。
<例1>
[4, 2, 1, 5, 3]
<例2>
['t', 'p', 'v', 'o', 'c']取りうる評価値があまりに離散的な場合(例えば [0, 1, 2] しか取り得ない等)、最適化ができないことがありますので、できるだけきめ細かな評価関数の設計を心がけましょう。
お手軽表示機能|show_pool_func=None, ‘bar’, ‘print’, ‘plot’, ‘(path)/’
vcoptには、GAの設定値や途中経過を可視化するためのお手軽表示機能が4種類用意されています。
show_pool_func=None の場合
設定値や途中経過は一切表示されません。計算はもっとも高速です。この指定は、vcoptを連続的に実行したり、多段階にネストして用いる場合に最適です。
show_pool_func=’bar’(デフォルト)
冒頭に設定値が表示され、途中経過が進捗バーで表示されます。計算は高速です。最小限の情報で十分な場合に最適です。
左端は目標評価値、+はエリートスコア、<はmean_gapです。<が+に追いつくと終了が近いです。
show_pool_func=’print’
冒頭に設定値が表示され、途中経過がprintで表示されます。計算は高速です。詳細な情報を見たい場合に最適です。
show_pool_func=’plot’
冒頭に設定値が表示され、途中経過がグラフで表示されます。pool_numが100より大きい場合、100体までのスコアが表示されます。計算は低速で、Noneと比較して3倍以上の時間がかかる場合があります。
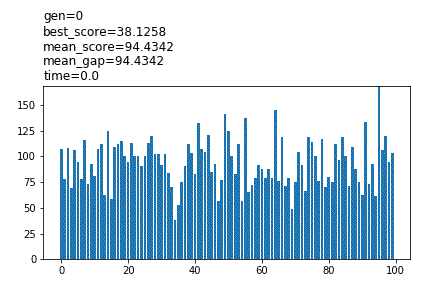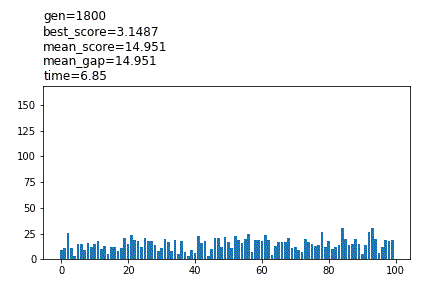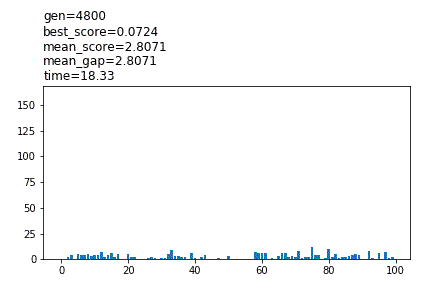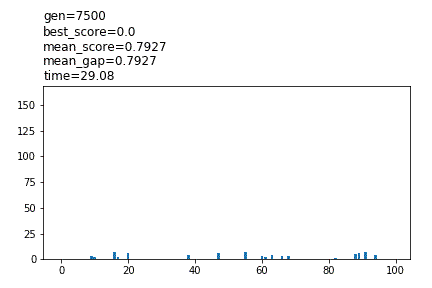
show_pool_func='(path)/’
コンソールには show_pool_func=’bar’ と同様の表示があると同時に、指定したフォルダ内に show_pool_func=’plot’ と同様のグラフが次々と保存されます。必ず ‘save/’ のように最後にスラッシュを付けてください。指定したフォルダが存在しない場合は自動的に作成されます。処理はもっとも低速です。
‘./’ を指定すると現フォルダに大量のグラフが保存されますのでご注意ください。また、’save’ のようにスラッシュを付け忘れると無効になります。
可視化関数の書き方|show_pool_func()
お手軽表示機能の他に、可視化関数を独自に定義し、show_pool_func=your_func のように渡すことで、GA中の個体群の様態や、最適値の推移を自由に見ることができます。
次のルールに従って書きます。
#poolの可視化
def show_pool_func(pool, **info):
#GA中の諸情報はinfoという辞書に格納されて渡されます
#これらを受け取って使用することができます
gen = info['gen'] #現在の世代
best_index = info['best_index'] #エリート個体のインデックス
best_score = info['best_score'] #エリート個体の評価値
mean_score = info['mean_score'] #個体群の平均評価値
mean_gap = info['mean_gap'] #目標値と評価値の差の絶対値平均
time = info['time'] #経過時間(秒)
#可視化
print(...)pool は次のような2次元配列です(ndarray型)。
[para,
para,
para,
para]pool[best_index] によりエリート個体の para が取得できます。
info[‘mean_score’] は目標値に対して片側から近づくタイプの問題で用いると便利です。一方で、info[‘mean_gap’] は目標値に対して両側から近づくタイプの問題で用いると便利です。
info[‘mean_score’] の使用例
個体群の評価値:[2.2, 2.4, 1.8, 1.6, 2.6, 2.0]
目標値:0.0
このような問題においては、mean_score=2.1 が参考になるでしょう。個体群の平均評価値が 2.1 であり、目標値まで 2.1 離れている事がわかります。
info[‘mean_gap’] の使用例
個体群の評価値:[9.2, 10.4, 10.8, 9.7, 9.3, 10.6]
目標値:10.0
このような問題においては、mean_score=10.0 よりも mean_gap=0.8 の方が参考になるでしょう。個体群が目標値まで平均 0.8 離れていることが分かります。
その他のTips
個体数の指定
デフォルトでは個体数が10n(nは次元数(=パラメータ数))となっているため、例えば800パラメータの最適化では個体数が8000となり、非常に時間がかかる場合があります。初期個体の評価中に次のようなカウントが表示されます。
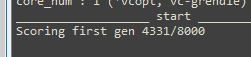
この進行があまりにも遅い場合は、pool_num=2000 のようにオプションを指定することができます。ただし、個体数を少なくすると大域解の探索力が低下する可能性があります。
逆に、個体数が少ないと感じた場合には増やすこともできます(離散性が高い問題、悪スケール性の問題において有効です)。
ライセンス
不明点はお気軽にお問い合わせください。
「vcopt を使ってみたブログを書く」○(著作権表示が必要)
「みんなも vcopt 使おうよ!」○
「vcopt を利用した商品を売る」○(著作権表示が必要)
「vcopt は私が作りました!」×
「vcopt のソースコードを修正する」×
「vcopt のラッパーを作成する」○(著作権表示が必要)
著作権表示
vcoptを使用してできた著作物(商品、論文、設計、プログラム、ブログ記事等)を不特定多数の人々が閲覧できる場所に公開する際には、vcoptが直接的または間接的に貢献したかにかかわらず、必ず著作権表示をしなければなりません。ただしこれは、
- vcoptがビネット&クラリティ(=ビネクラ)によって作られたことが明示されていること
- ビネット&クラリティのURL(https://vigne-cla.com/ または同/vcopt-tutorial/ または同/vcopt-specification/)が明示されていること
の両方が満たされていれば良いこととし、端的には
※vcoptはビネクラ(https://vigne-cla.com/)のソフトです
といった注釈を、十分な視認性を保って記載すれば良いこととします。また、二次的な営利利用にも本ライセンスを適用しなければなりません。例外として、vcoptの名称や機能のみを紹介した場合や、vcoptの名称や機能のみをデータベースの一要素として列挙した場合はこの限りではありません。
できること
個人利用、商用利用、創作物(商品、論文、設計、プログラム、ブログ記事等)の公開
禁止事項
ソースコードの修正・複製、pipを経由しないソースコードの配布、vcoptそのものやvcoptを利用した技術にかかる特許の出願・取得、トレードマークの主張、有償版(vc-grendel)のサブライセンス
免責事項
vcoptを用いたことで生じたいかなる損害の補償もしかねます。
更新予定
rcGA()終了条件の改善
rcGA()悪スケール性への対応強化
並列計算の改善
GPU計算への対応
tspGA()高速化
更新履歴
| 1.6.3 | 目標値が比較演算子を含む文字列に対応 設定値の表示を修正 rcGA()終了条件を修正 |
| 1.6.1 | dcGA()に独自の可視化関数を入力した際のエラーを修正 |
| 1.6.0 | 並列計算の実装(プレミアムパッケージ「vc-grendel」) 初期個体評価で目標値を達成した場合に対応 |
| 1.5.6 | 設定値の表示を実装 show_pool_func=’bar’の幅を調整、デフォルトに指定 show_pool_func='(path)/’オプションの実装 rcGA()の初期個体の多様性を向上 rcGA()を悪スケール性に対応(弱) rcGA()のパラメータ、終了条件を変更 入力形式のロバスト性を大幅に向上 ライセンス規定を更新 |
| 1.5.3 | 初期個体評価中のカウント機能を実装 |
| 1.5.2 | para_rangeがlist形式の入力に対応 rcGA(), dcGA()が1次元の入力に対応 |
| 1.5.1 | dcGA()の交叉法の変更 rcGA()の高速化 max_genオプションの実装 お手軽表示機能を実装(show_pool_func=’bar’, ‘print’, ‘plot’) |
| 1.5.0 | setGA()の実装 dcGA()の交叉法の変更 |
| 1.4.8 | int, float, strが混在した入力に対応 pool_numオプションの公式実装 mean_gapの実装 変数設計の見直し |
| 1.4.7 | pool_numオプションの非公式実装 |
| 1.4.5 | 進化規模均一化によるGAの安定化 |
| 1.4.3 | tspGA()の高速化 |
| 1.4.2 | dcGA(), rcGA()の実装 |
| 1.4.1 | opt2(), tspGA()の実装 |




