やること
k-means法を可視化していたら気がつきました。
細胞分裂後期っぽいなあ。
ということで今回はk-meansを応用して細胞分裂のシミュレーションを行います。
k-meansについて
k-means法のアルゴリズムと可視化についてはこちらの動画が分かりやすいです。
こちらも同様に分かりやすい。
細胞分裂について
Nicolas Minc, et al., Influence of Cell Geometry on Division-Plane Positioning, Cell 144(3), 414-426, 2011をご参照ください。アブストをGoogle翻訳にぶっ込み、図をサーっと見ます。
媒精したウニ卵をさまざまな形状のマイクロチャンバーに閉じ込め、第一卵割面の決まり方を実験と数理モデルで検討した論文です。
実行環境
WinPython3.6をおすすめしています。
Google Colaboratoryが利用可能です。

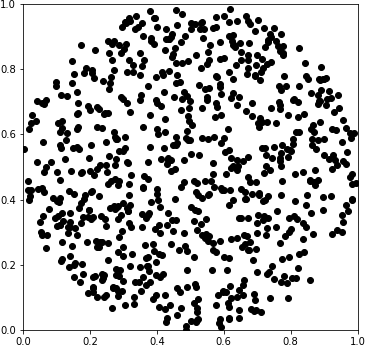
細胞を用意する
細胞に見立てた円状の点群を用意します。
import numpy as np
import numpy.random as nr
import matplotlib.pyplot as plt
#============================
# 設定(細胞の形状)
#============================
#円形
data = nr.rand(1000, 2)
mask = np.where(((data[:, 0] - 0.5)**2 + (data[:, 1] - 0.5)**2)**0.5 < 0.5)[0]
data = data[mask]
#============================
# 表示
#============================
plt.figure(figsize=(6, 6))
plt.scatter(data[:, 0], data[:, 1], c='k')
plt.xlim(0, 1); plt.ylim(0, 1)
plt.show(); plt.close(); print()
点群をk-meansする関数
sklearn.cluster.KMeans が使えればよいのですが、1ステップ毎に止めて可視化するような機能がなかったので、自分でk-meansを書きます。
ここでは「適当な重心」と「点群」を受け取って、k-meansを50ステップ進めてから「2つの重心の時系列データ」と「2つの点群の時系列データ」を返す関数を作りました。
#============================
# 重心と点群を受け取り、重心を2つに分裂させてk-meansする関数
#============================
def division(center, data):
#重心の分裂
center_1 = center + (nr.rand(2) * 0.01 - 0.005)
center_2 = center + (nr.rand(2) * 0.01 - 0.005)
#データ
center_time_1 = []
center_time_2 = []
data_time_1 = []
data_time_2 = []
#k-meansを50ステップ進める
for step in range(50):
print('step:{}'.format(step))
#2つの重心から各点までの距離計算
dist_1 = ((data[:, 0] - center_1[0])**2 + (data[:, 1] - center_1[1])**2)**0.5
dist_2 = ((data[:, 0] - center_2[0])**2 + (data[:, 1] - center_2[1])**2)**0.5
#重心1に属するかどうかのbool
bool_1 = dist_1 < dist_2
#各重心に属する点群
data_1 = data[bool_1, :]
data_2 = data[~bool_1, :]
#重心と所属点群を記録
center_time_1.append(center_1)
center_time_2.append(center_2)
data_time_1.append(data_1)
data_time_2.append(data_2)
#重心の更新(配下の点群の重心に移動)
center_1 = np.mean(data_1, axis=0)
center_2 = np.mean(data_2, axis=0)
#2つの重心と2つの点群の時系列データを返す(点群データのサイズはステップ毎に変わるのでndarrayにしないよう注意)
return center_time_1, center_time_2, data_time_1, data_time_2
#適当な初期重心
center = [nr.rand()*0.02 + 0.49, nr.rand()*0.02 + 0.49]
#分裂して2細胞期へ
center_time_1, center_time_2, data_time_1, data_time_2 = division(center, data)
print('len(center_time_1):\n{}'.format(len(center_time_1)))
print('len(data_time_1[0]):\n{}'.format(len(data_time_1[0])))
print('len(data_time_2[0]):\n{}'.format(len(data_time_2[0])))len(center_time_1):
50
len(data_time_1[0]):
381
len(data_time_2[0]):
382適当な初期重心と先ほどの点群を入れて、返り値のサイズを見てみました。重心1は(x, y)が50ステップ並んだ2次元配列です。1ステップ目に重心1に属した点群は381個、重心2に属した点群は382個でした。
k-meansを可視化する関数
何細胞期でも可視化できるように拡張性を意識して、可視化関数を作りました。
#============================
# 複数の重心、複数の点群の時系列データを受け取ってプロットする関数
#============================
def plot_frame(center_time_list, data_time_list):
#50ステップ表示
for step in range(50):
#表示
plt.figure(figsize=(6, 6))
#各細胞の重心とデータをプロット
for i in range(len(center_time_list)):
#この細胞、このステップの情報
center = center_time_list[i][step]
data = data_time_list[i][step]
#点群
plt.scatter(data[:, 0], data[:, 1])
#重心
plt.scatter(center[0], center[1], s=100)
#線(重い)
#for j in range(len(data)):
# plt.plot([data[j, 0], center[0]], [data[j, 1], center[1]], '-k', alpha=0.2)
plt.xlim(0, 1); plt.ylim(0, 1)
plt.title('step:{}'.format(i))
plt.show(); plt.close(); print()
#listに格納
center_time_list = [center_time_1, center_time_2]
data_time_list = [data_time_1, data_time_2]
#1→2細胞期を可視化
plot_frame(center_time_list, data_time_list)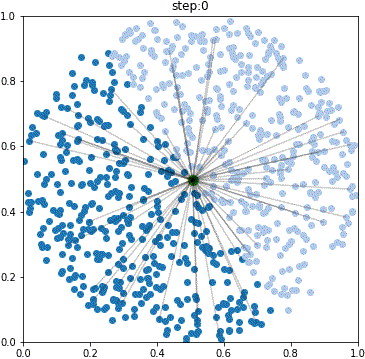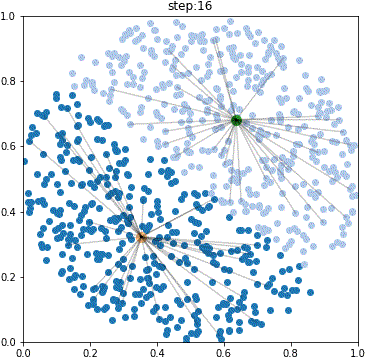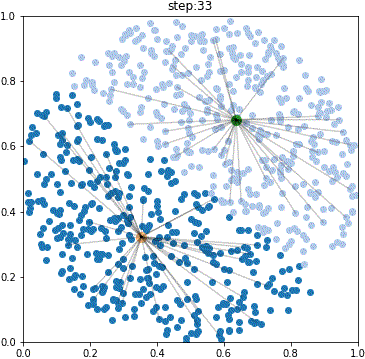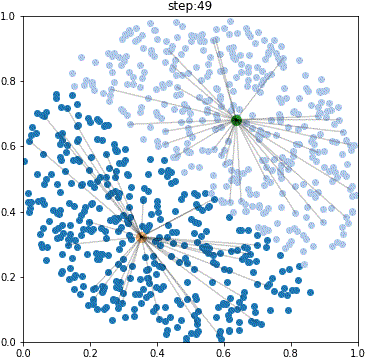
いい感じです。
コードの改良
要領が分かりました。まず、重心の移動が急すぎるので、移動の速さを30%くらいに落とします。
#重心の更新(配下の点群の重心に移動)
#center_1 = np.mean(data_1, axis=0)
#center_2 = np.mean(data_2, axis=0)
center_1 = center_1*0.7 + np.mean(data_1, axis=0)*0.3
center_2 = center_2*0.7 + np.mean(data_2, axis=0)*0.316細胞期までシミュレーションするコードです。
#適当な初期重心
center = [nr.rand()*0.02 + 0.49, nr.rand()*0.02 + 0.49]
#分裂して2細胞期へ
center_time_1, center_time_2, data_time_1, data_time_2 = division(center, data)
#listに格納
center_time_list = [center_time_1, center_time_2]
data_time_list = [data_time_1, data_time_2]
#1→2細胞期を可視化
plot_frame(center_time_list, data_time_list)
#分裂して4細胞期へ、listに格納
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[0][-1], data_time_list[0][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[1][-1], data_time_list[1][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
#2→4細胞期を可視化
plot_frame(center_time_list[-4:], data_time_list[-4:])
#分裂して8細胞期へ、listに格納
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[2][-1], data_time_list[2][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[3][-1], data_time_list[3][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[4][-1], data_time_list[4][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[5][-1], data_time_list[5][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
#4→8細胞期を可視化
plot_frame(center_time_list[-8:], data_time_list[-8:])
#分裂して16細胞期へ、listに格納
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[6][-1], data_time_list[6][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[7][-1], data_time_list[7][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[8][-1], data_time_list[8][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[9][-1], data_time_list[9][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[10][-1], data_time_list[10][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[11][-1], data_time_list[11][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[12][-1], data_time_list[12][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
center_time_1, center_time_2, data_time_1, data_time_2 = division(center_time_list[13][-1], data_time_list[13][-1])
center_time_list += [center_time_1, center_time_2]
data_time_list += [data_time_1, data_time_2]
#8→16細胞期を可視化
plot_frame(center_time_list[-16:], data_time_list[-16:])楕円形チャンバーで16細胞期まで
#============================
# 設定(細胞の形状)
#============================
#楕円
data = nr.rand(1000, 2)
mask = np.where(((data[:, 0] - 0.5)**2 + (data[:, 1] - 0.5)**2)**0.5 < 0.5)[0]
data = data[mask]
data[:, 1] = data[:, 1]*0.7 + 0.15疎な点群
密な点群
論文より比較用の画像を拝借します。
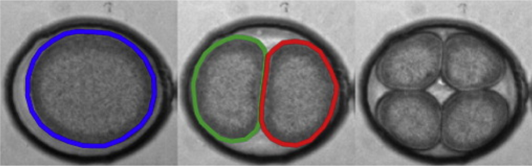
三角形チャンバーで16細胞期まで
#============================
# 設定(細胞の形状)
#============================
#三角
data = nr.rand(1000, 2)
mask = (data[:, 1] < 3.0*data[:, 0] - 0.5) * (data[:, 1] < -3.0*data[:, 0] + 2.5)
data = data[mask]疎な点群
密な点群
論文より比較用の画像を拝借します。
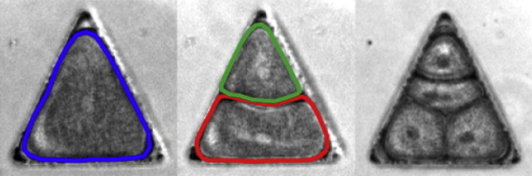
L字型チャンバーで16細胞期まで
#============================
# 設定(細胞の形状)
#============================
#L字
data = nr.rand(1000, 2)
mask = (data[:, 0] > 0.5) * (data[:, 1] > 0.5)
data = data[~mask]疎な点群
密な点群
楕円切り欠きチャンバーで16細胞期まで
#楕円切り欠き
data = nr.rand(1000, 2)
mask1 = np.where(((data[:, 0] - 0.5)**2 + (data[:, 1] - 0.5)**2)**0.5 < 0.5)[0]
data = data[mask1]
data[:, 1] = data[:, 1]*0.7 + 0.15
mask2 = (data[:, 1] < 4.0*data[:, 0] - 0.9) * (data[:, 1] < -4.0*data[:, 0] + 1.7)
data = data[~mask2]
mask3 = (data[:, 1] > 4.0*data[:, 0] - 0.7) * (data[:, 1] > -4.0*data[:, 0] + 1.9)
data = data[~mask3]疎な点群
密な点群
論文によれば、切り欠きのところで割れるわけではない。
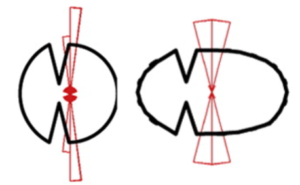
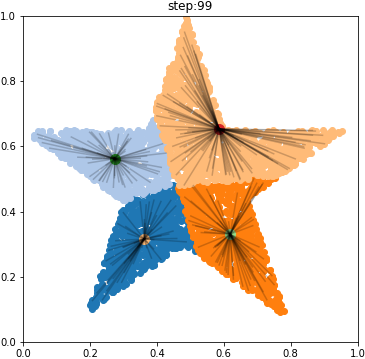
星型チャンバーで16細胞期まで
星型を作るのが面倒くさかったです。。
#星型
data = nr.rand(12000, 2)
mask1 = (data[:, 1] > 0.65) * (data[:, 1] > 3.08*data[:, 0] - 0.5)
data = data[~mask1]
mask2 = (data[:, 1] > 0.65) * (data[:, 1] > -3.08*data[:, 0] + 2.5)
data = data[~mask2]
mask3 = (data[:, 1] > 0.65) * (data[:, 1] > -3.08*data[:, 0] + 2.5)
data = data[~mask3]
mask4 = (data[:, 1] < -0.73*data[:, 0] + 0.65) * (data[:, 1] > 3.08*data[:, 0] - 0.5)
data = data[~mask4]
mask5 = (data[:, 1] < 0.73*data[:, 0] - 0.05) * (data[:, 1] > -3.08*data[:, 0] + 2.5)
data = data[~mask5]
mask6 = (data[:, 1] < 0.73*data[:, 0] - 0.05) * (data[:, 1] < -0.73*data[:, 0] + 0.65)
data = data[~mask6]疎な点群
密な点群
4細胞期に入るとき、大きい方の割れ方が興味深いですね。
考察・展望(レポートかな?)
点群の疎密による違い
- 疎なほど初期値依存性が高い(k-meansの初期値依存性が強く出る)
実際の細胞と似ている点
- 中心体の位置の決め方がそこそこ理論に則っている
- 収まりが悪いときに「ぐりん!」ってなる
- 変な切り欠きのところでは割れない
- (今回は2次元だがk-meansは任意の次元数でできるので3次元シミュレーションも可)
実際の細胞と異なる点
- 微小管は細胞内容物ではなく細胞膜を突っ張る
- 微小管の突っ張りや分裂溝形成による細胞形状の変化がない
- たぶんこのモデルでは極体放出を説明できない
ということで、次回があるとしたらよりリアルなシミュレーションに挑戦したいと思います。というか、k-meansなんて使わないで素直に微小管とアクチン線維のシミュレーションをすればいいんですが、それだと普通すぎて面白くないので…。

